
时间:2020-12-25浏览次数:4724
一、肺炎动物模型造模方法分类
肺炎动物模型的构建方法主要包括:细菌性肺炎、病毒性肺炎、真菌性肺炎、化学性肺炎及混合感染模型,不同方法模拟的病理机制与临床相关性存在差异。
二、细菌性肺炎模型
1. 常用方法
病原体:肺炎链球菌(Streptococcus pneumoniae)、金黄色葡萄球菌(Staphylococcus aureus)
造模方式:气管内滴注或雾化吸入活菌或灭活菌液。
2. 核心机制通路
TLR4/NF-κB信号通路:细菌脂多糖(LPS)通过TLR4激活NF-κB,诱导促炎因子(TNF-α、IL-6、IL-1β)释放。
NLRP3炎性小体通路:细菌毒素触发NLRP3炎性小体组装,促进IL-1β和IL-18成熟释放。
补体系统激活:C3a、C5a介导中性粒细胞募集。
3. 关键蛋白
TLR4:模式识别受体(PRR),识别LPS。
MyD88:TLR4下游衔接蛋白,激活NF-κB。
NLRP3/ASC/Caspase-1:炎性小体复合物关键组分。
HMGB1:晚期炎症介质,参与脓毒症相关肺损伤。
4. 相关临床研究
抗TLR4抗体:临床试验中用于抑制过度炎症反应(如Eritoran)。
IL-1β拮抗剂(Anakinra):用于脓毒症相关肺损伤的II期研究。
三、病毒性肺炎模型
1. 常用方法
病原体:流感病毒(如H1N1)、SARS-CoV-2(COVID-19模型)。
造模方式:鼻腔滴注病毒悬液。
2. 核心机制通路
RIG-I/MAVS通路:病毒RNA被RIG-I识别,激活MAVS信号,诱导I型干扰素(IFN-α/β)。
JAK-STAT通路:干扰素通过JAK-STAT信号增强抗病毒基因表达。
细胞焦亡(Pyroptosis):Gasdermin D介导的细胞死亡释放大量炎症因子。
3. 关键蛋白
RIG-I/MAVS:病毒RNA识别复合物。
IFNAR1/2:I型干扰素受体。
Gasdermin D:焦亡执行蛋白。
ACE2:SARS-CoV-2进入细胞的受体。
4. 相关临床研究
瑞德西韦(Remdesivir):靶向病毒RNA聚合酶,用于COVID-19治疗。
托珠单抗(抗IL-6R):抑制细胞因子风暴(COVID-19重症患者)。
四、真菌性肺炎模型
1. 常用方法
病原体:白色念珠菌(Candida albicans)、曲霉菌(Aspergillus fumigatus)。
造模方式:气管内注射真菌孢子或菌丝。
2. 核心机制通路
Dectin-1/Syk通路:识别真菌β-葡聚糖,激活Syk激酶,诱导ROS和炎性因子。
Th17免疫应答:IL-23/IL-17轴介导中性粒细胞募集。
NETosis:中性粒细胞释放胞外诱捕网(NETs)杀伤真菌。
3. 关键蛋白
Dectin-1:真菌模式识别受体。
IL-17A/IL-23:Th17细胞因子。
MPO(髓过氧化物酶):NETs形成的关键酶。
4. 相关临床研究
抗IL-17单抗(Secukinumab):临床试验中用于慢性肺曲霉病。
棘白菌素类(如卡泊芬净):靶向真菌细胞壁β-1,3-葡聚糖合成。
五、化学诱导肺炎模型
1. 常用方法
LPS(脂多糖)模型:气管内滴注LPS模拟急性肺损伤。
盐酸吸入模型:模拟胃酸误吸导致的吸入性肺炎。
2. 核心机制通路
TLR4/TRIF通路:LPS通过TLR4-TRIF激活IRF3,诱导IFN-β。
ROS/NLRP3通路:线粒体ROS激活NLRP3炎性小体。
MAPK通路:调控细胞凋亡与炎症反应。
3. 关键蛋白
TRIF:TLR4下游接头蛋白。
SOD2:线粒体抗氧化酶。
p38 MAPK:调控炎症细胞迁移。
4. 相关临床研究
NAC(N-乙酰半胱氨酸):通过抗氧化作用减轻肺损伤(ARDS患者)。
p38抑制剂(Losmapimod):II期临床试验用于COPD急性加重。
六、混合模型(细菌+病毒)
1. 常用方法
流感病毒继发细菌(如肺炎链球菌)感染模型。
2. 核心机制通路
IFN-γ抑制肺泡巨噬细胞功能:病毒抑制宿主抗菌免疫。
PD-1/PD-L1通路:T细胞耗竭加重继发感染。
3. 关键蛋白
PD-1:免疫检查点蛋白。
IFN-γ:抑制细菌清除的双刃剑效应。
4. 相关临床研究
PD-1抑制剂(如Pembrolizumab):探索用于重症病毒-细菌混合感染。
模型总结和对比
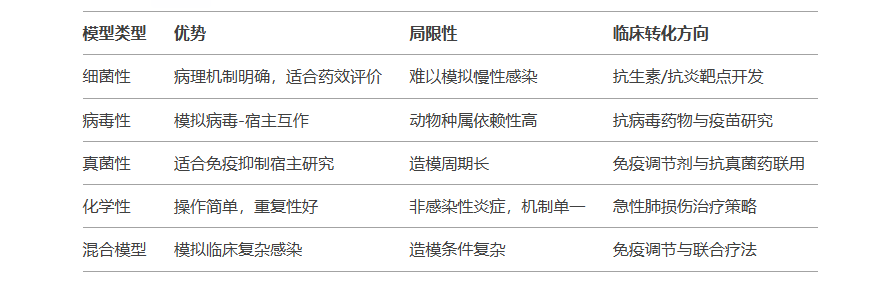
研究方向
模型选择:根据研究目的(急性/慢性、感染/免疫)选择合适模型。
多组学整合:结合转录组、蛋白组分析关键通路动态变化。
临床相关性验证:利用患者样本验证动物模型中的靶点(如IL-6、NLRP3)。
如需进一步探讨某类模型或分子机制,可提供具体方向深入分析。
